MOLEKULARBIOLOGISCHE TESTMETHODEN
Diese Methoden basieren auf einer Vervielfältigung der vorhandenen DNA mittels einer PCR (polymerase chain reaction).
Es sind qualitative Tests, die ein Gen oder sein Fehlen nachweisen. Damit wird der Genotyp untersucht und aus diesem kann in den meisten Fällen der Phänotyp abgeleitet werden.
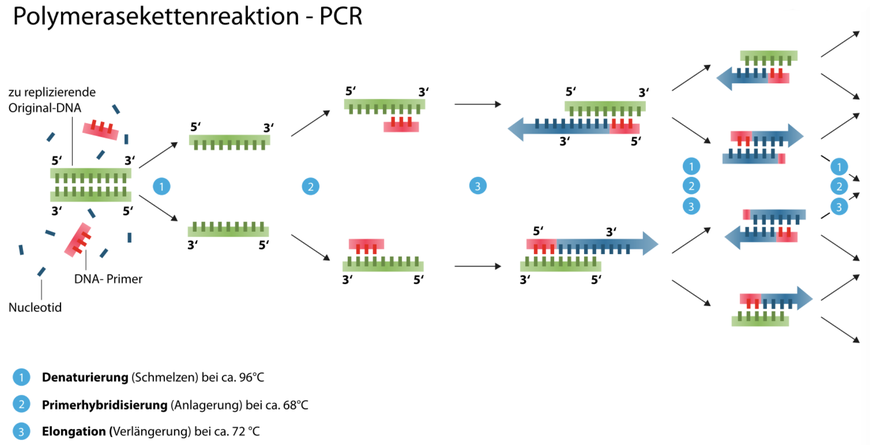
Enzoklop – Weitergabe unter gleichen Bedingungen 3.0 Deutschland
Sequence Specific Primer-PCR: (SSP)
hier werden zwei Primer verwendet, die die gesuchte Sequenz "umschliessen" - ein forward und ein backward Primer. Diese legen sich an die vorhandene DNA, wenn sie die Sequenz enthält und durch die PCR wird diese Sequenz so vervielfältigt, so sie sichtbar gemacht werden kann. Dafür wird eine kleine Menge des PCR-Produktes auf ein Gel aufgetragen und dieses unter Stromspannung gesetzt. Parallel läuft eine Größen-Referenzkontrolle. Sie wird auch Ladder genannt, weil sie auf dem Gel als ein Strichkode mit Strichen von unterschiedlichen Größen (Anzahl der Basenpaare - bp) aussieht. Unsere Sequenz hat eine uns bekannte Grösse und wenn wir auf dem Gel in der entsprechenden Höhe (Grösse) einen Strich sehen, dann wissen wir, dass diese Sequenz vorhanden ist.
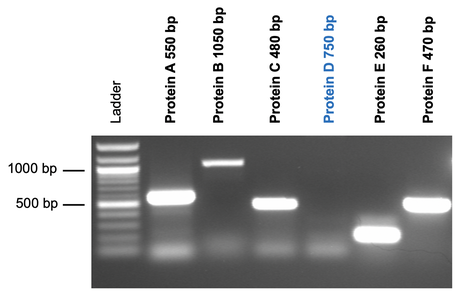
Auf diesem Foto sind PCR-Produkte von sechs Proteinen aufgetragen (A - F). Die DNA der Proteine A, B, C, E und F konnte nachgewiesen werden. Das Protein D war dagegen nicht im Untersuchungsmaterial vorhanden. Die Bande hätte bei 750 bp kommen sollen.
Die unterschiedliche Stärke der Banden liegt daran, dass die einzelnen Reaktionen in getrennten Ansätzen mit unterschiedlicher Ausgangs-DNA untersucht wurden. Ist die Menge an eingesetzter DNA unterschiedlich, führt es zu solchen Unterschieden in der Bandenstärke.
Diese Unterschiede können durch eine einheitliche Konzentration der eingesetzten DNA ausgeglichen werden. "Wünschenswert" wären Banden wie bei Protein B.
Sequenzspezifische Oligonukleotid - PCR: (SSO)
Auch hier wird zuerst eine PCR des uns interessierenden Lokus (z.B.: HLA-A) gemacht. Das Ergebnis der Reaktion wird in ein spezielles Näpfchen pipettiert. Am Boden dieses Näpfchens sind kurze Gensequenzen angebracht, die als Sonden dienen, wenn sie eine Komplementärsequenz in der Probe finden. Diese Bindung wird durch eine Farbreaktion sichtbar gemacht, Die Auswertung der SSO erfolgt über eine anbieterspezifische Software. Die bunten Reaktionen am Boden des Reagenzgefäßes werden fotografiert und das Muster durch die Software ausgewertet.
Das Testprinzip ist hier nochmal dargestellt:
(Die Zeichnung stammt von der BAG-Diagnostics Webseite:)
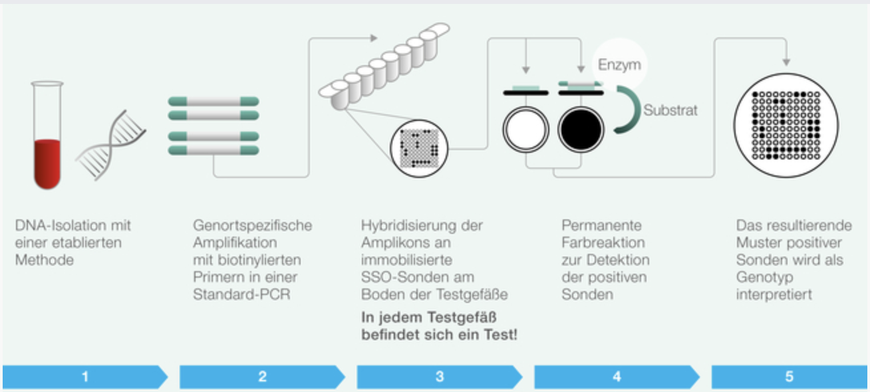

Der hier abgebildete runde Boden des Reaktionsgefäßes ist in Wirklichkeit nur ca. 7mm im Durchmesser. Die Software, in die das Muster der Reaktionen eingelesen wird, kann auch manuell nachkorrigiert werden. Wenn zum Beispiel eine Reaktion zu schwach erscheint und von der Software doch als positiv gewertet wurde. Lässt man das Ergebnis ohne Änderung, kann unter Umständen kein Ergebnis erzielt werden. Wird diese eine Reaktion manuell als negativ bewertet, bekommt man ein Ergebnis.
SEQUENZIERUNG - SBT (SEQUENCE BASED TYPING):
Bei der Sequenzierung wird die Basen-Sequenz der DNA ermittelt.
1975 hat Frederick Sanger mit seinem Team ein enzymatisches Analyseverfahren zur Bestimmung der Basen-Reihenfolge in der DNA entwickelt - die Kettenabbruchmethode. 1980 bekam Sanger zusammen mit Gilbert dafür den Nobelpreis in Chemie.
Erst mit dieser Methode ist es möglich geworden, alle HLA-Gene auf Allelebene zu bestimmen - 4 Digit.
Das ermöglichte auch eine Suche nach HLA-identen KM-Spendern.
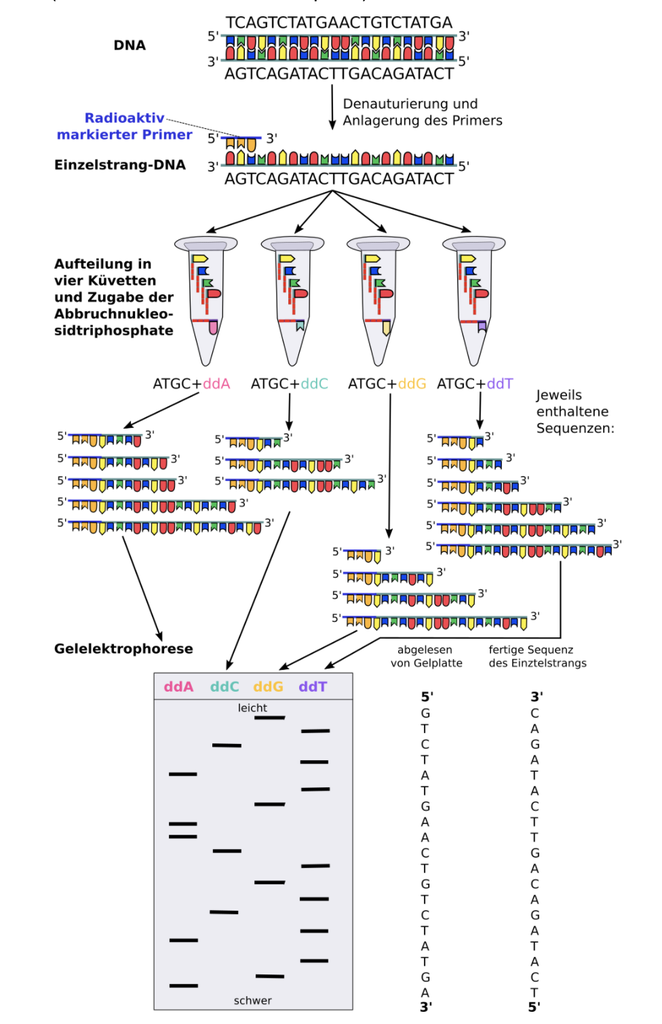
Sequenzierung nach Sanger:
Die Methode wird auch Kettenabbruchmethode bezeichnet.
Für die Durchführung braucht man
- Einsträngige DNA
- DNA-Polymerase
- Radioaktiv markierte Primer
- dNTPs - Desoxynukleotid-Triphosphate (dATP, dTTP, dCTP, dGTP) - radioaktiv markiert
- ddNTPs - (Di-Desoxynukleotid-Triphosphate - ddATP, ddTTP, ddCTP, ddGTP) - diesen fehlt Hydroxygruppe, so dass sie kein weiteres Nukleotid mehr binden kann. Sie werden auch Abbruchnukleotide genannt. Auch sie sind radioaktiv markiert.
Wenn man jetzt die Reaktion startet, werden die denaturierten DNA, DNA-Polymerase, Primer, dNTPs (dATP, dTTP, dCTP, dGTP). Es werden vier Tubes (Reagenzröhrchen) benötigt, die sich nur in dem zusätzlichen Abbruchnukleotid (ddNTP) unterscheiden.
Die Reaktion im Gefäß mit ddATP beginnt mit dem Primer und bricht immer dann ab, wenn an der Stelle von dATP zufällig ddATP eingebaut wurde. Dadurch werden viele unterschiedlich lange DNA-Stücke generiert.
Die fertigen Produkte werden auf ein Gel aufgetragen. Je kürzer ein Stück ist, umso weiter läuft es im Gel.
Aus einem Tube enden alle DNA Stücke immer mit dem gleichen Nukleotid - dem Abbruchnukleotid in diesem Tube.
Die nach Größe aufgetrennten DNA-Stücke für alle 4 Tubes entsprechen der Reihenfolge der Basen - diese Information entnehme ich der Zuordnung zum jeweiligen Abbruchnukleotid.
serologische testmethoden
Mittels Antikörper-Antigen-Reaktionen können im CDC Klasse I Antigene bestimmt werden und auch die Verträglichkeitsprobe zwischen Spenderzellen und Empfängerserum durchgeführt werden.
Zu den serologischen Methoden gehören auch die ELISA-Bestimmung der HLA-Antikörper.
Ein wesentlich sensitiver Test ist der Luminex-Screening-Test, der auf Polystyren-Beads basiert.
Ebenfalls Bead-basiert ist die Antikörperspezifikation mittels Single-Antigen-Beads.
CDC (complement dependent cytotoxicity):
Loci A, B und C können serologisch mittels CDC-Tests (complement-dependent cytotoxicity) bestimmt. Es werden Platten verwendet, die mit mono- oder polyspezifischen Antikörpern gegen HLA Klasse I Antigene beschichtet sind. Es sind 96-Well Platten, wo jedes Näpfchen mit definierten Antikörpern beschichtet ist. Nun werden isolierte Lymphozyten des Patienten in die Näpfchen pipettiert und zusammen mit Komplement inkubiert - daher die Bezeichnung "Komplement-abhängige-Zytotoxizität". Zum Schluss werden die Zellen mit einem Farbstoff gefärbt, der tote Zellen rot färbt. Zellen, die nicht durch die Antikörper und das Komplement angegriffen wurden, diese bleiben grün. Jetzt kann anhand der Reaktionsmuster der HLA-Typ Klasse I abgelesen werden. Wenn z.B.: Zellen im Näpfchen mit Anti-A2 und Anti-A24 rot gefärbt sind, heißt es, dass sich genau diese Antigene auf der Oberfläche der Zellen befinden. Also ist der HLA-A Typ des Patienten A2, A24. Das gleiche gilt für den B-Lokus und C-Lokus. Kommt ein Antigen homozygot (es ist nur ein Antigen nachweisbar) oder der HLA-Typ lässt sich serologisch nicht eindeutig bestimmen, werden molekularbiologische Methoden eingesetzt.
Mit der gleichen Methode können zytotoxische HLA-Antikörper nachgewiesen werden. Eingesetzt wird es bei der Verträglichkeitsprobe vor Nieren und Pankreas-Transplantationen. Diese Antikörper können im schlimmsten Fall zu einer sofortigen Abstossungsreaktion führen, die bereits während der OP einsetzt. Bei allen anderen Organen kann die CDC-Verträglichkeitsprobe (auch Kreuzprobe oder Kreuzversuch, KV genannt) auch nach der Transplantation durchgeführt werden.
Bei dem Test werden die Lymphozyten der Spender mit dem Serum des Patienten in Anwesenheit von Komplement inkubiert. Sind im Serum des Patienten HLA-AK vorhanden, die das Komplement aktivieren können, werden dadurch die Lymphozyten zerstört. Zum Schluss kommt eine Farblösung dazu, die nur tote Zellen rot anfärbt. Intakte Zellen bleiben grün. Die Reaktion wird als positiv gewertet, wenn 20% der im Mikroskop sichtbaren Zellen, rot sind. Bei einer positiven Kreuzprobe darf eine Niere oder Pankreas nicht transplantiert werden.
Sollte bei einem anderen Organ die nachträgliche Kreuzprobe positiv sein, muss die Immunsuppression dementsprechend angepasst werden. Die oben beschriebene akute Abstossungsreaktion ist dabei weniger ein Problem.
LUMINEX Screening:
Bei dieser Technologie werden Polystyren-Beads mit mehreren HLA-Antigenen Klasse I oder Klasse II beladen. Die Beads werden mit dem Patienten-Serum inkubiert, wobei die Antikörper gegen HLA-Antigene an die Beads binden. Mit einem weiteren, Fluoreszenz-markierten Antikörper, der gegen humane Antikörper gerichtet ist, kann die Beladung der einzelnen Beads gemessen werden. Obwohl die Antigen-Spezifitäten der einzelnen Beads bekannt sind, können einzelne Antikörper damit nicht definiert werden. Aber der Test ist wesentlich sensitiver und weniger störanfällig wie der LAT-Elisa.
LUMINEX Single Antigen:
Es gibt Situationen, wo die Spezifität der HLA-Antikörper von Bedeutung ist. Vor Transplantationen - um die HLA-Antigene zu definieren, welche der Patient nicht bekommen darf und nach Transplantationen, um die Immunantwort des Patienten gegen das transplantierte Organ zu monitieren.
Hat das transplantierte Organ ein HLA-A2 Antigen auf der Oberfläche und der Patient bildet nach der Transplantation einen Antikörper gegen A2, muss die Immunsuppression angepasst und der Patient auf eine Abstossung überwacht werden.
Der Test funktioniert auf dem gleichen Prinzip wie Luminex Screening. Es sind wieder Polystyren-Beads, die aber diesmal mit einzelnen HLA-Antigen-Molekülen beschichtet sind. Dort binden dann die HLA-Antikörper der Patienten und sie werden mit einem Fluoreszenz-markierten Anti-human-IgG Antikörper sichtbar gemacht. Jedes Bead hat seine eigene Fluoreszenz, die gemessen wird, um das Bead zu erkennen. Gleichzeitig wird die Fluoreszenz der gebundenen Antikörper gemessen. So kann zugeordnet werden, welches Bead wie stark fluoresziert. Diese Fluoreszenz entspricht theoretisch der Menge an gebundenen Antikörpern, die in weiterer Folge der Menge der Antikörper im Patientenserum entspricht.
Die übliche Art diese Antikörper zu interpretieren ist die Angabe von MFI (Mean Fluorescence Intensity). Dadurch wird der nicht ganz korrekte Eindruck erweckt, als wäre der Test eindeutig quantitativ. Die Bindung der Antikörper an die Beads wird durch weitere Faktoren beeinflusst, die nicht ohne weiteres beurteilt werden können. Zum Beispiel sind Antigene, die auf den Beads fixiert sind, nicht in ihrer "natürlichen Umgebung". Dadurch kann sich die dreidimensionale Form des Antigens verändern. Und das kann dazu führen, dass ein Antikörper entweder schwächer oder stärker bindet. Und auch die Beladung der einzelnen Beads mit Antigenen ist nicht für alle Spezifitäten gleich hoch. Vergleiche ich jetzt zwei unterschiedliche Beads miteinander, die voll beladen sind, erscheint eines schwächer, weil nicht mehr Antikörper daran binden konnte.
Es ist ein sehr wertvoller Test, der aber nur einen Teil der Diagnostik bilden darf.
Ausserdem wie alle empfindlichen Methoden hat auch diese ihre Nachteile. Es werden viele Antikörper erfasst, die im CDC-Test nicht zu sehen sind. Das bedeutet, dass sie offensichtlich nicht zytotoxisch wirken. Die klinische Relevanz dieser Antikörper beim einzelnen Patienten ist nicht immer klar. Aber die Studien zeigen, dass Patienten mit starken MFI-Werten bei donorspezifischen Antikörpern, ein höheres Risiko für eine antikörpervermittelte Abstossung (AMR) haben.
Zuletzt bearbeitet am 19.08.2021